Tocantins colhe 9,1 milhões de toneladas de grãos em 2024/25
O volume estabelece um novo recorde na série histórica do Estado; soja milho e arroz são destaques
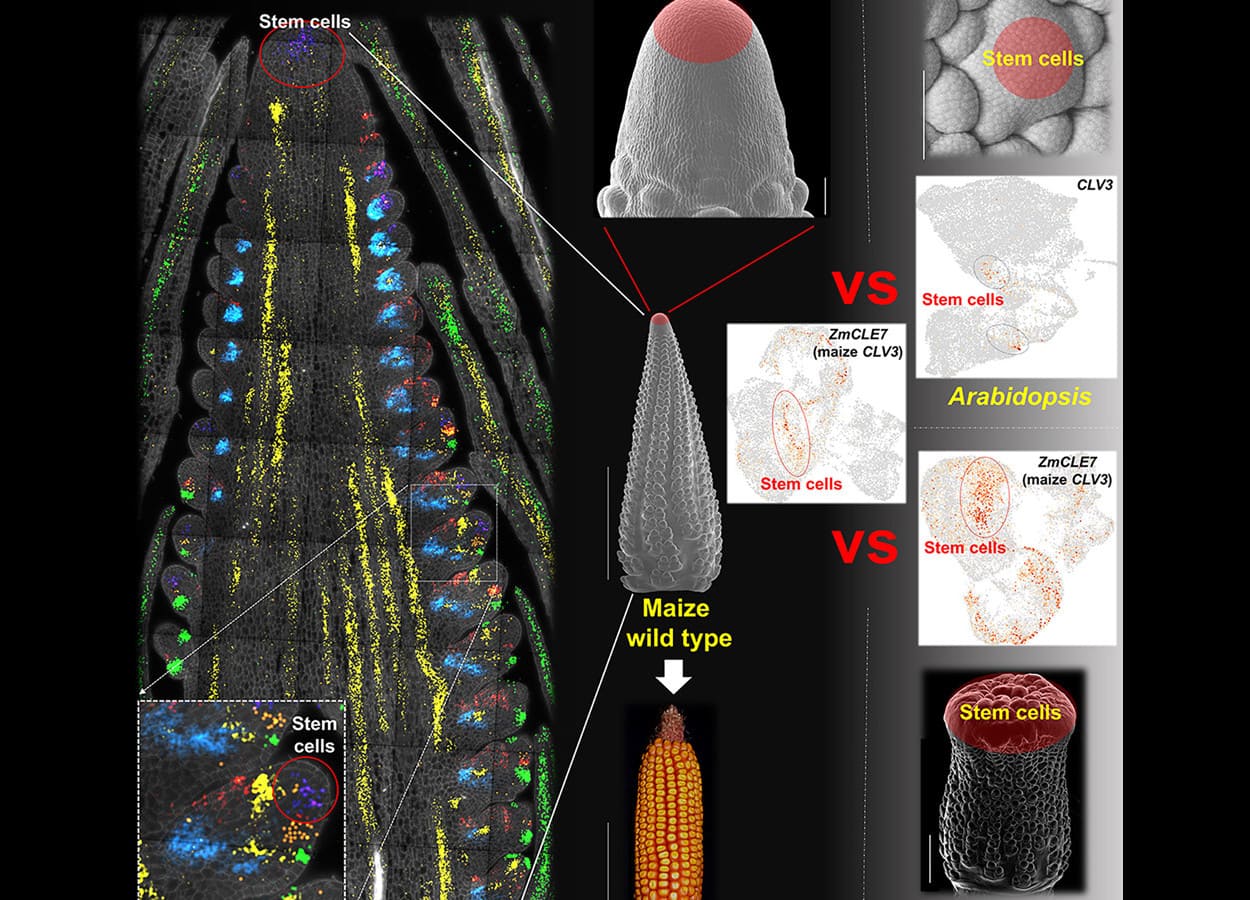
A pesquisadores do Laboratório Cold Spring Harbor (CSHL), nos Estados Unidos, mapearam dois reguladores clássicos de células-tronco em milhares de células da parte aérea de milho e Arabidopsis sp. O estudo revelou novos genes reguladores e ligou alguns deles a características agronômicas importantes.
O trabalho, liderado pelo professor David Jackson, usou uma técnica chamada sequenciamento de RNA de célula única. A equipe analisou tecidos com alto teor de células-tronco, identificou a expressão de milhares de genes e produziu um “atlas” genético acessível a outros pesquisadores.
Segundo Jackson, os dados podem guiar avanços genéticos por uma década e ajudar no desenvolvimento de cultivos mais produtivos e resilientes.
As células-tronco vegetais localizam-se em regiões chamadas meristemas. Lá, genes como CLAVATA3 (CLV3) e WUSCHEL (WUS) controlam a proliferação celular. Entender como esses genes atuam permite manipular o crescimento e a arquitetura das plantas.
No entanto, análises anteriores enfrentavam dificuldades para isolar células expressando CLV3 e WUS, devido à baixa quantidade e fragilidade dessas células.
A equipe de Jackson superou esse obstáculo com microdissecção precisa e uso de microfluídica. Cada célula coletada teve seu RNA convertido em DNA, etiquetado e sequenciado. O grupo recuperou cerca de 5 mil células expressando CLV3 e mil células expressando WUS.
A técnica possibilitou a identificação de centenas de genes preferencialmente expressos em células-tronco de milho e Arabidopsis. Muitos deles se mostraram conservados entre as espécies, indicando funções importantes ao longo da evolução vegetal.
Entre os novos genes identificados, destacam-se famílias de proteínas ligadoras de RNA e genes quinases relacionados a metabolismo de açúcares, alguns com funções em homeostase hormonal. Esses genes, segundo os autores, agem de forma redundante para controlar o desenvolvimento dos meristemas.
A análise de variantes genéticas naturais em diferentes linhagens de milho indicou que alguns desses genes reguladores se associam a traços produtivos, como número de fileiras de grãos e comprimento da espiga. As informações abrem caminhos para engenharia racional de cultivares.
Entre os genes validados, os SERBP, semelhantes a reguladores presentes em humanos, mostraram papel crucial no desenvolvimento da parte aérea das plantas. Cerca de 70% dos genes conservados entre milho e Arabidopsis codificam proteínas ribossomais, o que sugere que a especialização dos ribossomos pode influenciar a diferenciação de células-tronco vegetais.
A equipe também analisou mutantes com proliferação excessiva de células-tronco. Os pesquisadores encontraram diferenças na expressão de genes ligados a vias de regulação hormonal, como os genes ADK, que influenciam a produção de citocininas (hormônios envolvidos na formação de novos órgãos). Parte desses genes parece atuar por uma via alternativa à clássica via LOG, expandindo o entendimento sobre o controle hormonal em plantas.
Genes como PFKB, também identificados, possuem homólogos bacterianos que respondem à hipóxia (condição que regula a pluripotência em células-tronco humanas). A descoberta sugere que fatores ambientais, como oxigenação, podem influenciar o comportamento das células-tronco em plantas por mecanismos ainda pouco explorados.
O estudo forneceu um modelo para identificar genes reguladores em outras espécies vegetais. A base de dados gerada já está disponível publicamente e pode orientar melhoramentos genéticos com foco em produtividade, arquitetura e resiliência.
Outras informações em doi.org/10.1016/j.devcel.2025.07.024

Receba por e-mail as últimas notícias sobre agricultura