Frio recorde em São Paulo preocupa setor agropecuário
Climatempo aponta que a mínima deve alcançar 4°C nesta quarta-feira (25)

A resistência genética da lagarta-do-cartucho (Spodoptera frugiperda) a toxinas de Bacillus thuringiensis (Bt) acaba de ganhar contornos mais definidos. Um grupo internacional de pesquisadores utilizou a técnica CRISPR/Cas9 para eliminar seletivamente genes candidatos ao papel de receptores de proteínas inseticidas. Os resultados apontam o gene SfABCC2 como peça central no enigma da resistência de Spodoptera frugiperda às toxinas Cry1Fa e Cry1Ab.
A identificação ocorreu após a criação de linhagens geneticamente modificadas com deleções nos genes SfABCC2, SfABCC3, SfCad1 e SfAPN1. Apenas a interrupção de SfABCC2 — isoladamente ou em conjunto com SfABCC3 — conferiu resistência significativa às toxinas testadas. As demais deleções não alteraram a suscetibilidade do inseto.
Os dados revelam uma resistência superior a 3.300 vezes contra Cry1Fa e mais de 450 vezes contra Cry1Ab, comparadas à linhagem suscetível YJ-19. Essas toxinas, derivadas da bactéria Bacillus thuringiensis, compõem o arsenal das lavouras transgênicas no controle de pragas. A descoberta reforça o alerta: o gene SfABCC2 tornou-se o flanco exposto dessa tecnologia.
A resistência exibiu herança autossômica recessiva no caso da Cry1Fa e recessiva incompleta para Cry1Ab. Essa distinção indica que o mesmo receptor, ao interagir com diferentes toxinas, pode gerar padrões distintos de dominância genética. O achado sugere uma complexidade funcional ainda não compreendida, e impõe novos desafios ao manejo de resistência.
Pesquisadores injetaram o complexo Cas9/sgRNA em ovos recém-postos da linhagem YJ-19. Essa linhagem, coletada em 2019 na província de Yunnan, China, nunca havia sido exposta a inseticidas ou toxinas Bt. A edição visou deletar longos fragmentos dos genes de interesse. A seleção das mutações envolveu PCR, sequenciamento e cruzamentos sucessivos até o isolamento de linhagens homozigotas.
Cinco linhagens foram obtidas: SfABCC2-KO, SfABCC3-KO, SfCad1-KO, SfAPN1-KO e uma dupla deleção SfC2/C3-KO. Bioensaios com Cry1Fa, Cry1Ab e Vip3Aa determinaram os níveis de resistência. Apenas SfABCC2-KO e SfC2/C3-KO sobreviveram massivamente às toxinas Cry1, demonstrando que o gene SfABCC2 é o elo essencial da toxicidade.
A linhagem SfC2/C3-KO mostrou resistência equivalente à de SfABCC2-KO, enquanto SfABCC3-KO sozinha não conferiu resistência. A conclusão: SfABCC3 não é funcionalmente redundante com SfABCC2 em S. frugiperda. Isso contrasta com outros insetos, como Helicoverpa armigera, em que é necessária a deleção dupla para manifestação da resistência.
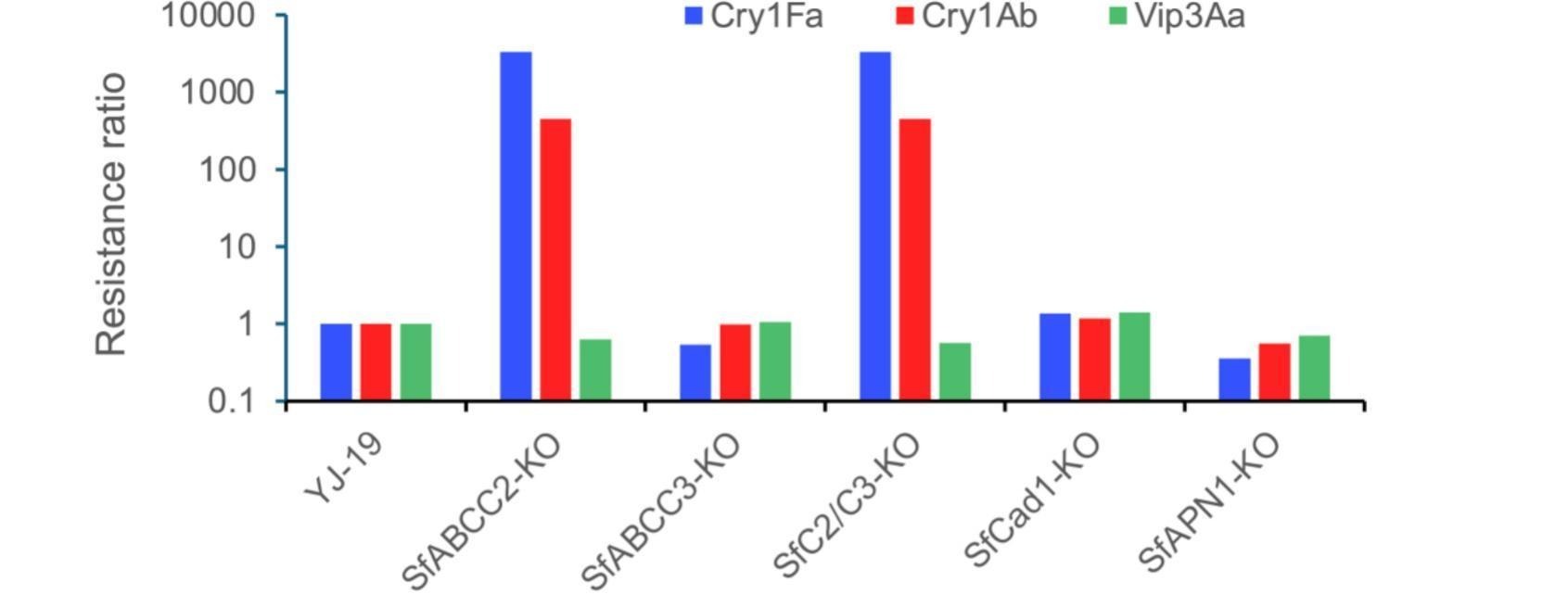
Outros dois genes, SfCad1 e SfAPN1, mostraram-se irrelevantes para resistência. Linhagens com esses genes deletados mantiveram susceptibilidade idêntica à linhagem controle. Resultados anteriores já apontavam função limitada dessas proteínas como receptores em Spodoptera.
Curiosamente, nenhuma das cinco linhagens apresentou resistência à toxina Vip3Aa. Isso confirma a especificidade dos genes deletados como receptores exclusivos de Cry1Fa e Cry1Ab. Também sinaliza que Vip3Aa continua eficaz, mesmo em populações com mutações em SfABCC2.
Experimentos adicionais determinaram a dominância genética da resistência. Cruzamentos entre linhagens resistentes e suscetíveis geraram F1s incapazes de sobreviver a Cry1Fa, mas com sobrevivência parcial a Cry1Ab. Isso demonstra resistência recessiva para Cry1Fa e incompletamente recessiva para Cry1Ab.
Testes de ligação genética comprovaram a associação entre resistência e deleção em SfABCC2. Larvas heterozigotas sucumbiram às toxinas. Apenas homozigotos resistiram, confirmando a ligação total entre genótipo e fenótipo resistente.
A resistência observada impõe restrições ao uso isolado das toxinas Cry1Fa e Cry1Ab. A forte resistência cruzada inviabiliza sua aplicação em pirâmides de genes Bt. A permanência de linhagens resistentes sem perda de aptidão sugere que a resistência pode se fixar em populações naturais sem custo evolutivo.
Na escala molecular, sete mutações já foram identificadas em SfABCC2 em populações de campo. Incluem três substituições de aminoácidos e quatro terminações prematuras. Algumas mutações reduzem a expressão do gene. Outras inativam a proteína receptora. Essas alterações ilustram a plasticidade genética da lagarta.
Mais informações em doi.org/10.1016/j.pestbp.2025.106526

Receba por e-mail as últimas notícias sobre agricultura