Greening: Defesa Agropecuária de Goiás detecta plantas contaminadas em dois municípios do Estado
Agrodefesa está promovendo medidas para controle de HLB no Estado após a identificação dos dois focos
Pesquisadores do Laboratório Nacional de Ames do Departamento de Energia dos Estados Unidos realizaram investigação sobre quitinases presentes em milho e arroz. Queriam entender melhor o papel dessas enzimas como indicadoras de estresse em plantas e seu potencial no combate a ataques fúngicos. Suas descobertas sugerem que essas quitinases podem ser recursos valiosos para monitorar a saúde das culturas.
Um dos maiores desafios para a produtividade agrícola é a presença, em grandes quantidades, de patógenos como fungos, bactérias e vírus. Os fungos, por exemplo, causam uma perda de rendimento de 35% a 40% nas culturas de milho e arroz anualmente.
Marit Nilsen-Hamilton, cientista do Ames Lab e líder da equipe de pesquisa, explicou haver “muito interesse em quitinases no mundo das plantas devido à sua ocorrência geral e seu envolvimento na proteção das plantas contra ataques fúngicos”.
Sua pesquisa foca principalmente na rizosfera, uma zona estreita de solo ao redor das raízes das plantas onde ocorre intensa atividade microbiana. As raízes liberam várias substâncias no solo, incluindo açúcares e quitinases, além de polímeros que mantêm tudo unido.
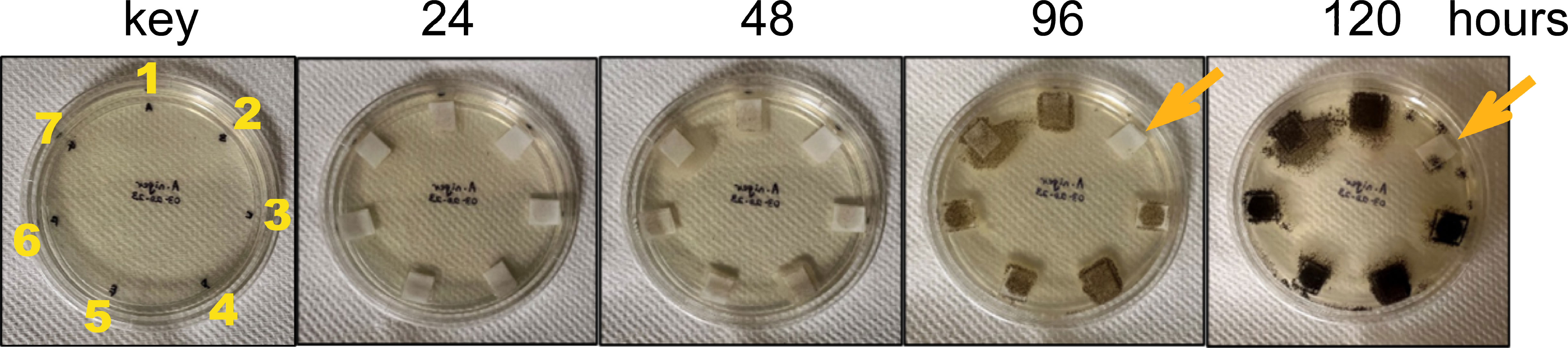
O trabalho inicial da equipe foi entender as características funcionais e moleculares das quitinases. O objetivo era determinar se elas são boas indicadoras de estresse relacionado a fungos nas plantas e se são capazes de matar fungos. Os pesquisadores descobriram que uma das quitinases foi eficaz em matar o fungo Aspergillus niger, que pode causar mofo-negro em várias culturas agrícolas.
Além de caracterizar as quitinases, a equipe explorou se elas poderiam ser bons receptores para aptâmeros. Eles são "ácidos nucleicos que se comportam como anticorpos, mas você não precisa de um animal para produzi-los", explicou Nilsen-Hamilton.
Os aptâmeros podem ser usados para detectar quitinases e potencialmente ativar certas quitinases secretadas em resposta a infecções fúngicas. A quitinase do arroz se mostrou um alvo inadequado para aptâmeros devido à repulsão entre cargas negativas, mas a quitinase do milho se revelou um bom receptor de aptâmeros.
A caracterização dessas quitinases é uma contribuição importante para a capacidade da equipe de detectar essas proteínas liberadas pelas plantas, permitindo que os agricultores intervenham precocemente contra infecções fúngicas.
Artigo publicado pelos cientistas a partir da pesquisa recebeu o seguinte resumo:
"As quitinases degradam a quitina, um homopolímero linear de resíduos de N-acetil-D-glucosamina (GlcNAc) ligados a β-1,4 encontrados nas paredes celulares de fungos e nos exoesqueletos de artrópodes. Eles são secretados pelas raízes na rizosfera, um ambiente complexo e dinâmico onde ocorre intensa troca de nutrientes entre plantas e micróbios.
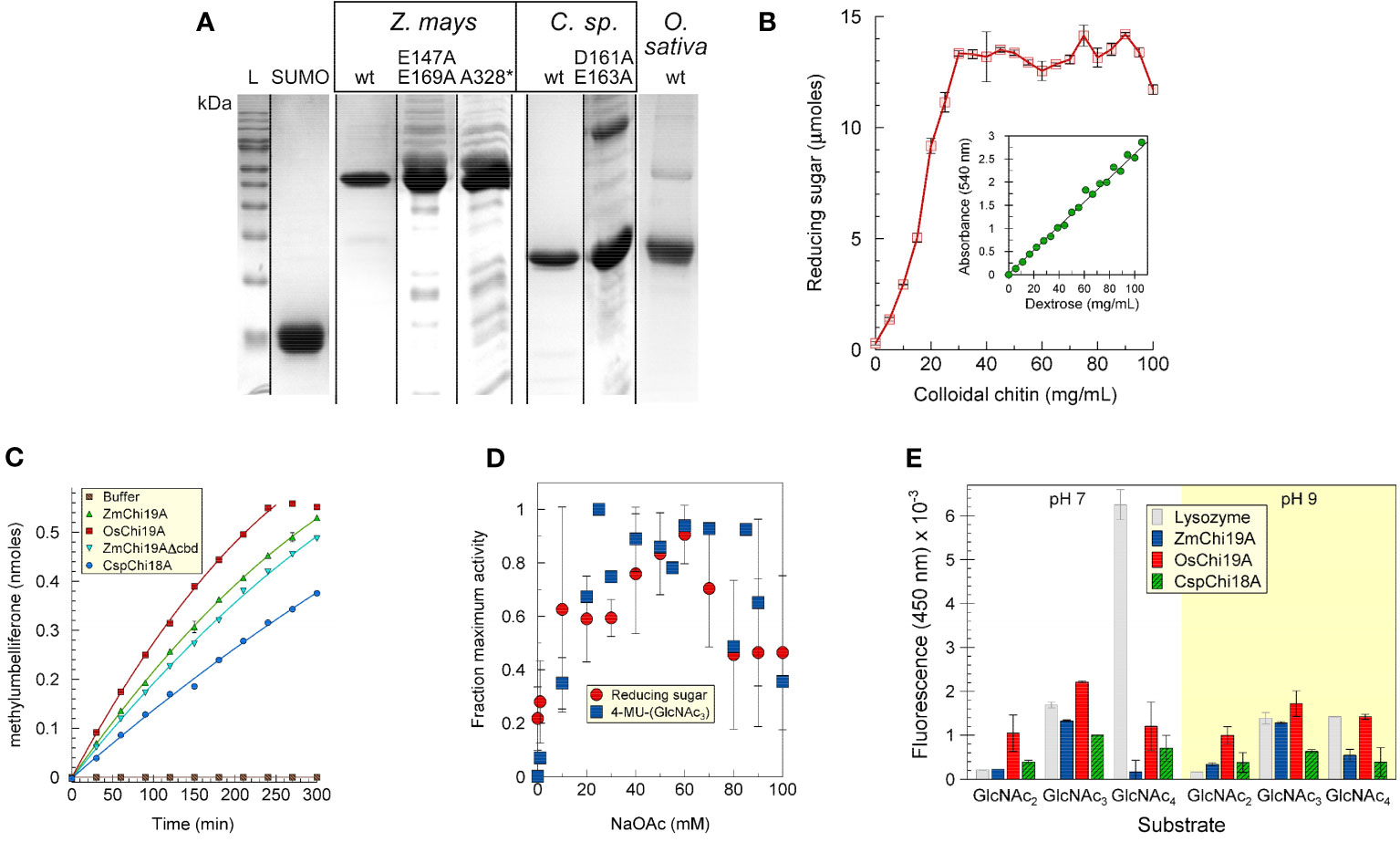
Aqui modelamos, expressamos, purificamos e caracterizamos quitinases de raiz de Zea mays e Oryza sativa, e a quitinase de uma bactéria simbiótica, Chitinophaga oryzae 1303, por suas atividades com quitina, di-, tri- e tetrassacarídeos e Aspergillus niger, com o objetivo de determinar seu(s) papel(s) na rizosfera e compreender melhor os mecanismos moleculares subjacentes às interações planta-micróbio. Mostramos que a endoquitinase básica de Zea mays (ZmChi19A) e a quitinase de Oryza sativa (OsChi19A) são da família da quitinase GH19. A quitinase Chitinophaga oryzae 1303 (CspCh18A) pertence à família GH18. As três enzimas têm valores KM aparentes semelhantes de (20-40 µM) para o substrato 4-MU-GlcNAc3. Eles variam em seu pH e temperatura ótimos com atividade OsChi19A ideal entre pH 5–7 e 30–40°C, enquanto as atividades ZmChi19A e CspCh18A foram ótimas em pH 7-9 e 50–60°C. A modelagem e a mutação dirigida ao local de ZmChi19A identificaram a fenda catalítica e os resíduos ativos E147 e E169 estrategicamente posicionados a ~ 8,6Å um do outro na proteína dobrada. A clivagem de 4-MU-GlcNAc3 não foi afetada pela ausência do CBD, mas diminuiu na ausência do domínio C-terminal flexível. No entanto, ao contrário do substrato solúvel, o CBD e o domínio C-terminal flexível recentemente identificado foram vitais para inibir o crescimento de Aspergillus niger.
Os resultados são consistentes com o envolvimento das quitinases vegetais na defesa contra patógenos como fungos que possuem exoesqueletos de quitina. Em resumo, caracterizamos as características funcionais e os domínios estruturais necessários para a atividade de duas quitinases de raízes de plantas que se acredita estarem envolvidas na defesa das plantas e uma quitinase bacteriana que, juntamente com as quitinases de plantas, pode participar na reciclagem de nutrientes na rizosfera.”
Mais informações podem ser obtidas em doi.org/10.3389/fpls.2024.1344142
Informações sobre quitinases: enzyme.expasy.org/EC/3.2.1.14
Informações sobre aptâmeros: en.wikipedia.org/wiki/Aptamer

Receba por e-mail as últimas notícias sobre agricultura